@agpwhy
2021-04-14T03:31:31.000000Z
字数 1492
阅读 326
王胖的生信笔记3:智人的优势-使用工具
因为找不到太好的R Markdown编辑平台,就在这里分享吧。因为这个平台不支持评论,有问题的话欢迎电子邮件shengmingwangjat@qq.com联系。电脑上阅读,效果更佳哦。
和各类猛兽飞禽比,人类的体力特质在大部分方面都是弱势的。那人类是如何猎获到力量/速度/灵巧远甚于自己的目标的呢?答案是团队合作&工具的使用。团队合作并不是此篇的重点,今天的重点是讲讲工具的使用。就以R Studio里的工具包使用为例。

简单提两句,R包就是R里面的工具包。因为我们不是专业计算机专业出身,写代码的功底没这么强。相比较写代码非常溜的那批人,我们作为入门选手也想完成一些远甚于自己基本能力的一些目标。这个时候就需要使用一些雷锋们贡献的免费工具包为我所用。只要学会安装和加载工具包,调用工具包内的函数,灵活修改一些参数就做到调用一个函数顶自己写几十上百行的作用了。
安装R包的几种姿势
请打开R Studio。
安装R包第一种方式
在左上方的代码区使用这段代码进行工具包gglot2的安装。
install.packages("ggplot2")
(注意括号内需要带上英语的引号)
安装R包的第二种方式
如果大家平时用R多的话,会发现配置环境时经常会出现这样的代码BiocManager::install(c("GenomicFeatures", "AnnotationDbi"))
相比较install.packages()来说,BiocManager的优势是不同的包间会有互相的依赖(比如不少的包都会对ggplot有依赖,即需要安装ggplot2包才能成功安装一些包),由于有些包会不断更新换代。依赖有时候一个包可能会和依赖的包之间有冲突。
所以当你确定使用一个BiocManager的版本后,所有用这个版本的BiocManager安装的包就不会因为包各自版本的原因而冲突。
install.packages("BiocManager")
BiocManager::install("ggplot2")
这样之后再用BiocManager::install()安装任何包就可以保证大部分的稳定了(我水平不够,有些时候包的安装对我来说是玄学问题,不敢说百分百)。
安装R包的第三种方式
今天大家使用以上两种已经能满足99%的基本需求了。然而凡事有例外。
比如说有时候你想用一个R包的开发版本,抑或者看到了一个还没有上架CRAN或者BiocManager的工具包想安装。那这个时候有一些其他的方式了。这里介绍一个需要一定前置设置的。(请大家自行搜索如何正常使用github)
devtools::install_github()
比如Rayshader这个包就可以使用这个包安装,可以完成一些非常炫的3D图。

阅读工具说明书
当然再好的工具包还要会使用才能有效。
library(ggplot2)
data(iris)
library(ggpubr)
library(ggthemes)
ggscatter(iris, x = "Petal.Length", y = "Sepal.Length", color = "Species") + theme_base()
比如用以上这段就可以画出这样的图了(里面没有安装过的包请自行参照上文安装)
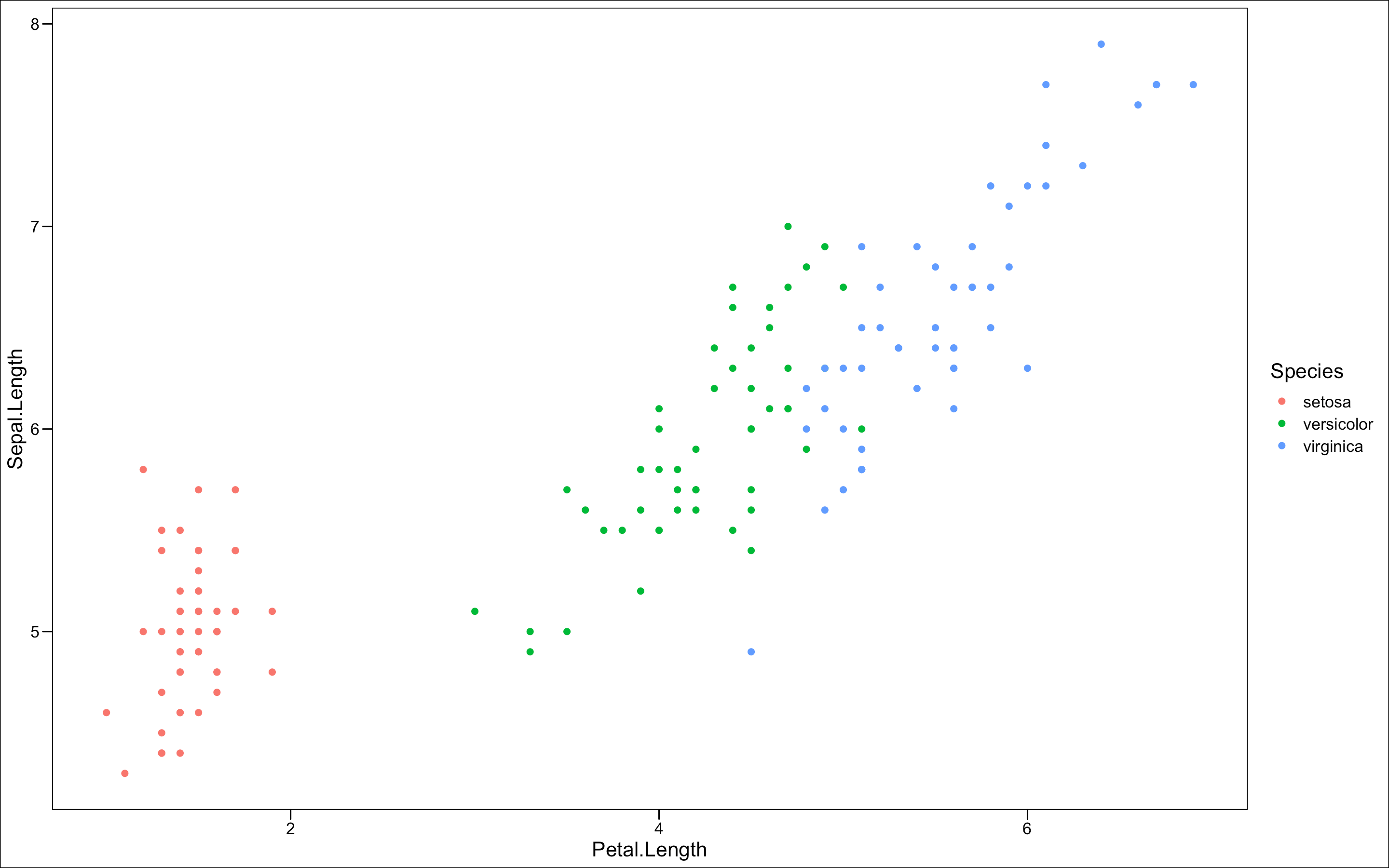
这比自己用ggplot2画再调参数快多了。
但是即使是知道一个函数可以完成一个目标,怎么灵活使用并调整参数呢?
当然可以百度。但是更直接的方式是直接?ggscatter
会有非常详细的说明出现(对于大部分包来说)
最后留一个小的问题。如何卸载已经安装的包?
