@agpwhy
2022-05-08T12:57:00.000000Z
字数 1296
阅读 346
王胖的生信笔记第四十一期:森林图
什么是森林图
其实之前“水”Meta大潮的时候,应该大家对于森林图有印象。
简单来说,就是横坐标表示一个参数,每个平行的线段代表一个研究对象(Meta里是一篇文章),然后线段展示这个研究对象这个参数的置信区间。几个研究对象合并后,可以加一个菱形或其他图形再表示这么多研究对象加权后这个参数的置信区间。
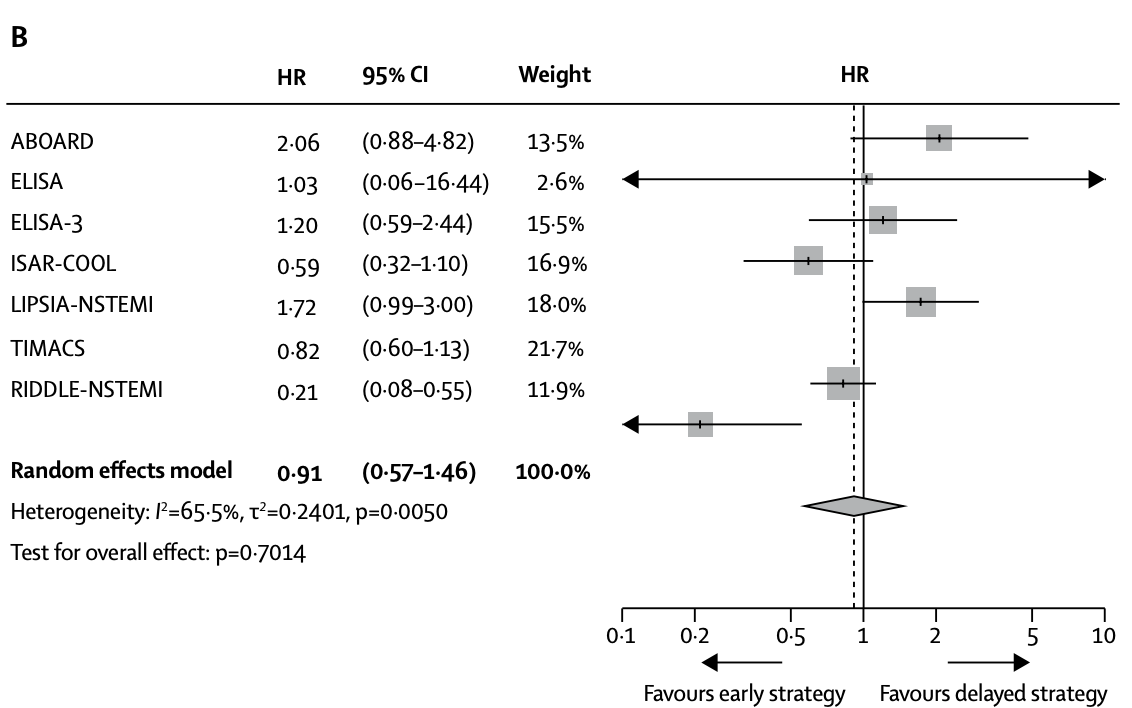
比如这个Lancet上的Meta文章

里这张森林图,讲的是不同研究non-fatal myocardial infarction的HR
Meta分析外还有其他森林图应用场景
对各种回归模型(regression models)比如CoxPH(Cox比例风险回归模型)和GLMs(广义线性模型),森林图也是重要的可视化手段。
Cox模型和临床医学生最相关的就是生存分析。这里就来介绍一下王诗翔大神推出的regport包。
这个包是基于R6类对象的,和Seurat那种S4有点不一样,不过这里先不做赘述(我也讲不清,后面学清楚了再写个笔记吧)。
准备
install.packages(c("broom.helpers","forestploter","see"))remotes::install_github("ShixiangWang/regport")library(regport)library(survival)lung = survival::lung #加载Surival包自带的Lung数据head(lung)
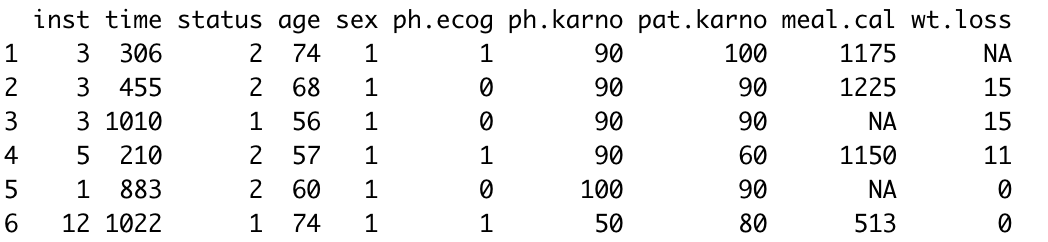
然后准备回归模型
lung$sex = factor(lung$sex)model = REGModel$new(lung,recipe = list(x = c("age", "sex"),y = c("time", "status")))# 另一种建立回归模型方法model = REGModel$new(lung,recipe = Surv(time, status) ~ age + sex)

线性回归模型我也学的不够好,我有机会写个小笔记写一下。
出图开始
有点丑的做图
model$plot()

这个肯定不行,但是这个包可以两行改成好看的
model$get_forest_data()
model$plot_forest()
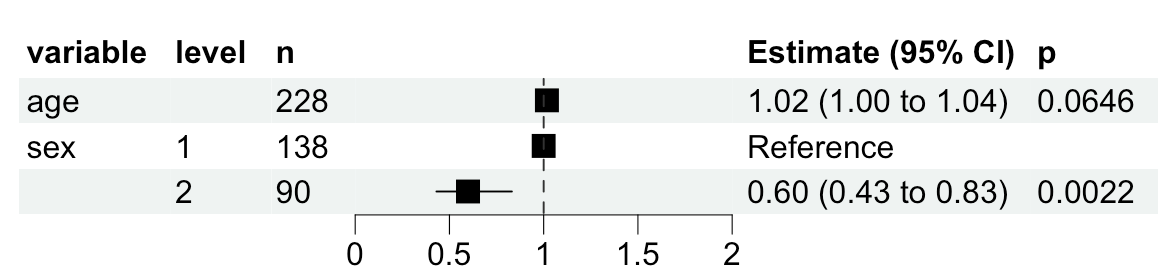
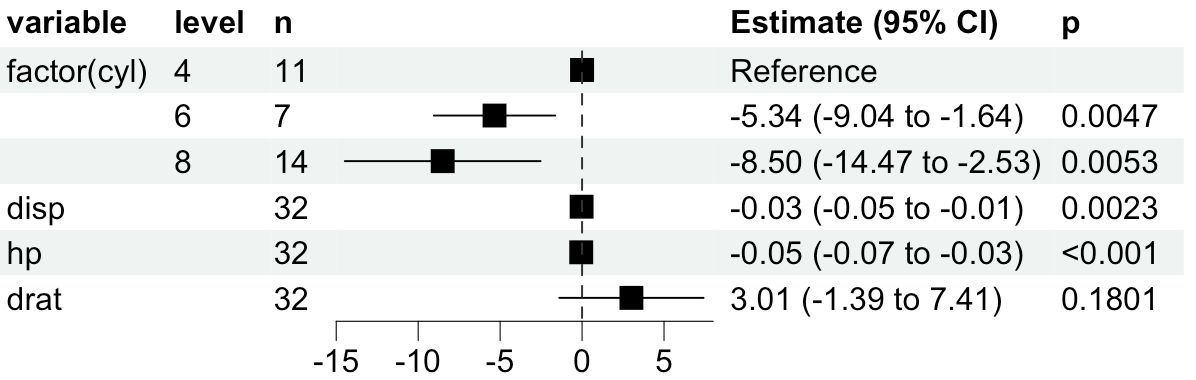
多加一些
ml <- REGModelList$new(data = mtcars,y = "mpg",x = c("factor(cyl)", colnames(mtcars)[3:5]),covars = c(colnames(mtcars)[8:9], "factor(gear)"))ml$build(f = "gaussian")ml$plot_forest(ref_line = 0, xlim = c(-15, 8))
就出来这样分层,带P的图
从院内宿舍开始自闭(3.7)到现在已经两个月了。我确实有很多自己给自己施加的压力,希望上海加油吧。我争取每周一篇再写到50篇吧。
