@agpwhy
2021-11-05T11:58:04.000000Z
字数 1940
阅读 318
王胖的生信笔记第二十五期—芯片的后续分析
下周会比较忙,我先提前更一下下周的内容。
上次说到了怎么下载数据和简单指控。现在我们默认已经完成了之前的步骤,对于GSE158850这个数据我们已经做了前面的步骤。那后面呢?
提取临床信息
同时在看几个数据,为了防止之后我自己搞不清哪个数据,就直接按照GSE好来命名了。
pdata=pData(GSE158850[[1]])
可以看下pdata的内容
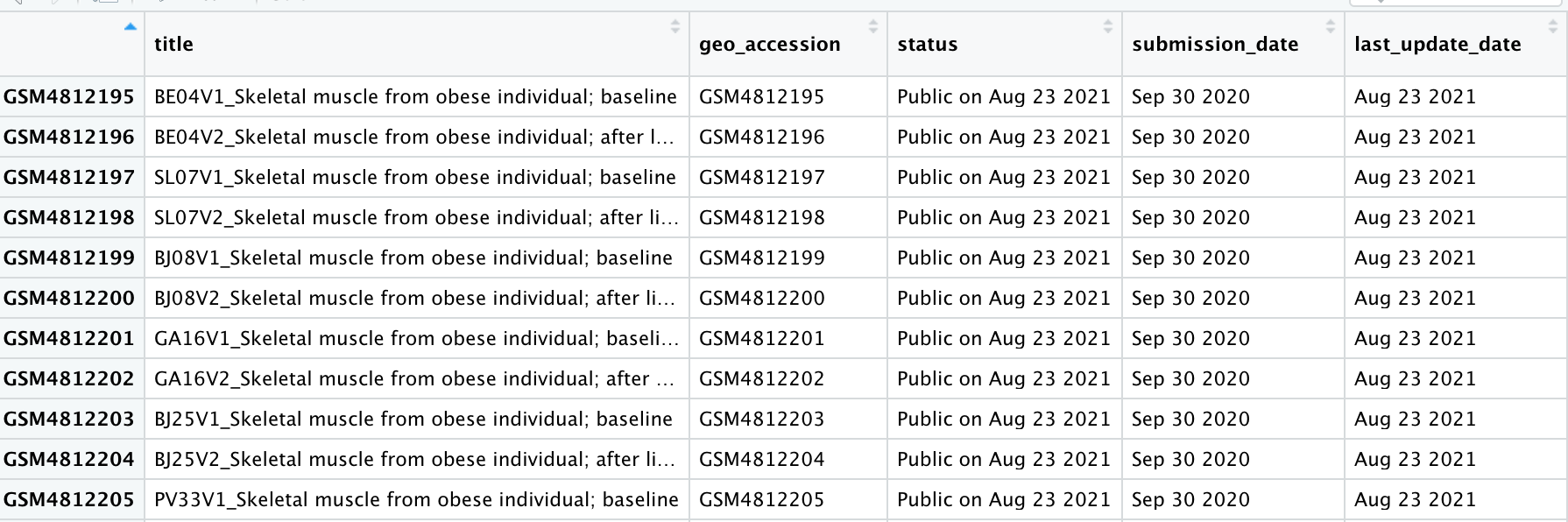
实际上只是内容的一小部分

后面呢,有两种操作方式,简单傻瓜一点的可以这样。
group_list<-case_when((str_detect(pdata$characteristics_ch2.2, "Young")) &(str_detect(pdata$time point:ch2, "baseline")) ~ "Young_Baseline",(str_detect(pdata$characteristics_ch2.2,"Young")&str_detect(pdata$time point:ch2,"intervention"))~"Young_Intervention",(str_detect(pdata$characteristics_ch2.2,"Elderly")&str_detect(pdata$time point:ch2,"baseline"))~"Elderly_Baseline",(str_detect(pdata$characteristics_ch2.2,"Elderly")&str_detect(pdata$time point:ch2,"intervention"))~"Elderly_Intervention")
或者学习下stringr包,
用聪明一点的方式。
individuals=factor(unlist(lapply(pdata$title,function(x) strsplit(as.character(x),"V")[[1]][1])))
treatment=unlist(lapply(pdata$title,function(x) strsplit(as.character(x),";")[[1]][2]))
treatment=factor(treatment,levels=c(" baseline"," after lifestyle intervention"))
这里一定要自己去跑一下,看一下stringr的介绍。光抄写这个没法推广到其他应用场景的。
进一步分析
这里就提到一个很简单的包了,tinyarray。
library(tinyarray)
dcp = multi_deg_all(ex,group_list,ids,adjust = F)
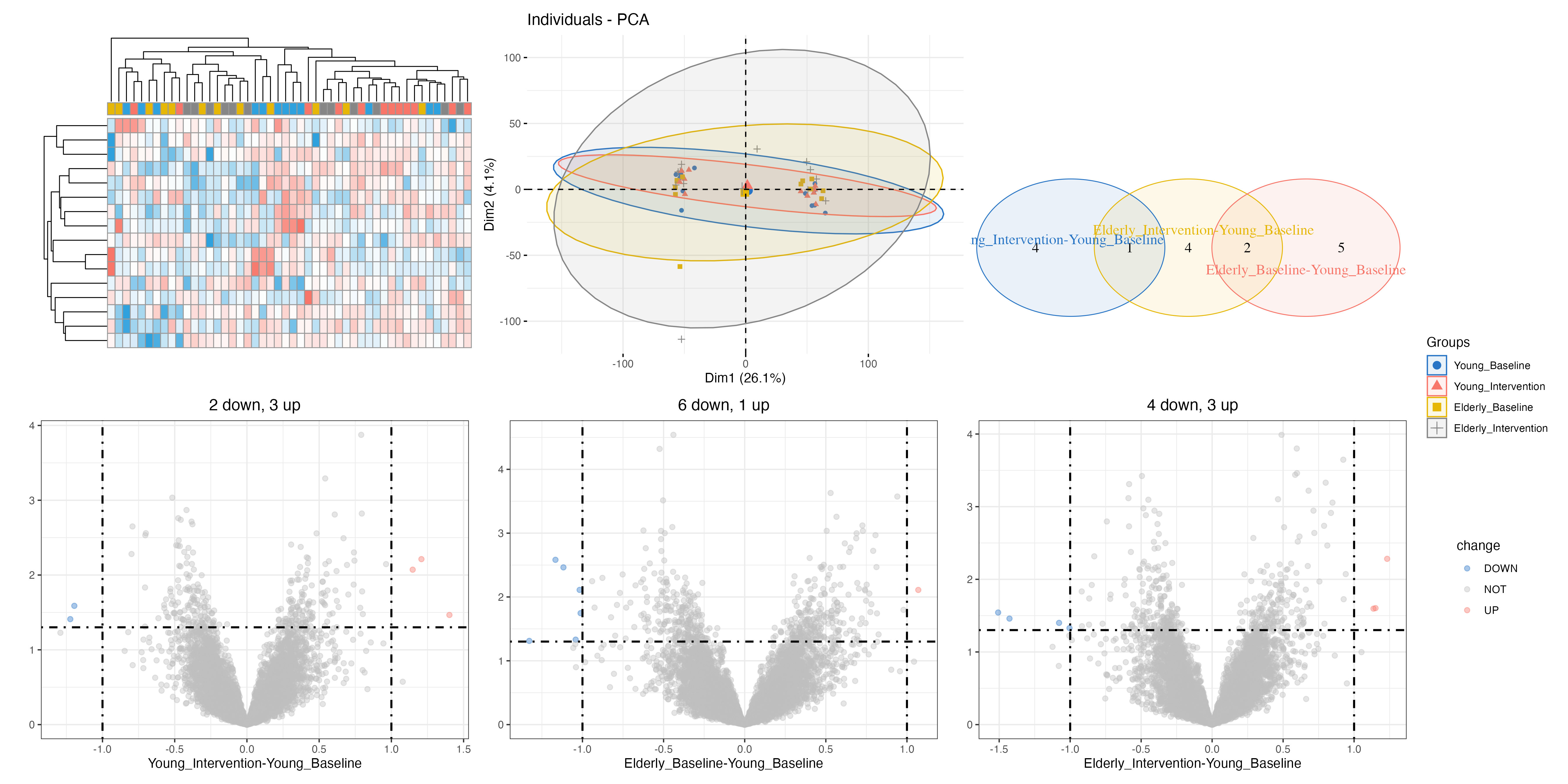
直接就把热图,火山图,韦恩图,PCA都搞定了。
【当然这里就跳过上次教程那里的id置换了,直接替你搞定了,否则会报错】
你会说我的天呐,这怎么这么好,那为啥还要学习其他方式呢?
因为有些东西简单的包没法完成,或者暂时没开放接口。
比如这个数据是可以进行配对比较的(同一个病人有干预前后的数据)
那怎么办呢?那要回到传统的方式。不过这里需要大家自己去学习一下什么事设计矩阵,什么是对比矩阵。老实说我这部分内容也不是在大学的统计学课堂里学的(这部分其实我觉得可以加入医学统计学里面,因为确实对于理解,写代码分析一些数据非常有用),而是在外面学一个影像学数据处理的时候,老师顺便教的。说实话教的蛮好的,可惜ppt是人的知识产权,而且这个东西光看ppt也没用,需要自己去理解的。我确实数学功底不好,大概是懂了,但是叫我给大家上课有点虚。总之一句话,如果想更好的掌握一些统计学比较和代码,还是要学一下基础的知识好一些。我这里就单纯展示如何实现配对检验了。
先来个不用配对的
design=model.matrix(~treatment)
fit=lmFit(exprSet,design)
fit=eBayes(fit)
TvsB_NonPaired <- topTable(fit,coef='treatment after lifestyle intervention',n=Inf,adjust='BH')
再来个要配对的
design=model.matrix(~individuals+treatment)
fit=lmFit(exprSet,design)
fit=eBayes(fit)
TvsB_Paired <- topTable(fit,coef='treatment after lifestyle intervention',n=Inf,adjust='BH')
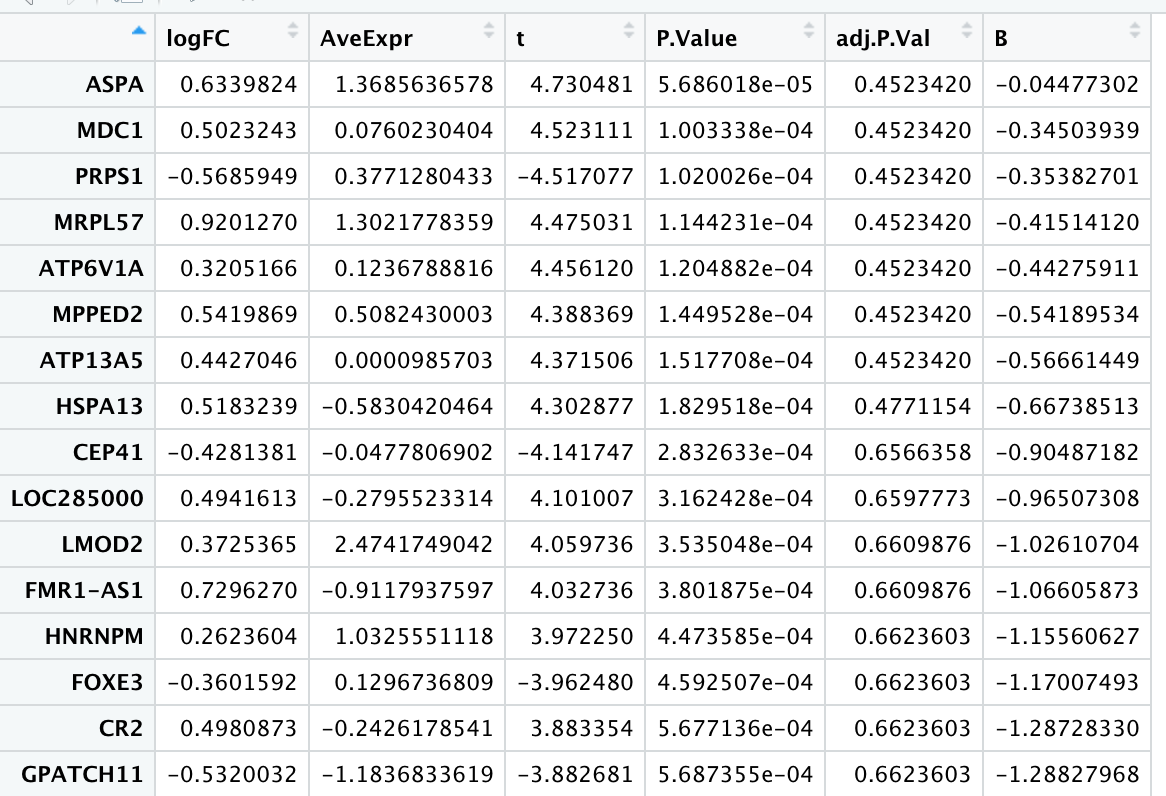
后续做pca啊,火山图啊, 热图啊,其实都是可以自己搞定的。感兴趣的人多我们一期期写笔记。
