@agpwhy
2022-02-07T08:36:56.000000Z
字数 1855
阅读 413
用R做网络图:王胖的生信笔记第36期
其实最常见的网络图做法,是用cytoscape。这里大家可以自己找可靠的教程,b站上也有,直接搜就可以。我觉得讲的蛮清楚的,使用也相对傻瓜。
但有时候我们做的数据就是在R里面的,不想搞这么一套来回的折腾,直接在R里面做行不行呢?
答案当然是可以的。而且还能够直接生成网页自己玩儿。
【动图不多放了,大家知道这是动态的就好】
使用Visnetwork
https://datastorm-open.github.io/visNetwork/
这是个巴黎理工大学一个团队做的,https://www.datastorm.fr/,我觉得还是R里一个很酷的工具。
其实和cytoscape一样,你需要node(节点)和edge(连接节点的线)。
library(visNetwork)nodes <- data.frame(id = 1:3)edges <- data.frame(from = c(1,2), to = c(1,3))visNetwork(nodes, edges, width = "100%")
这就是刚刚一个最简单的例子。
其实这个工具很强大。我这里就不露怯了,大家可以自行探索。
结合自己的数据-比如转录调控网络
比如大家拿一堆基因算出来的调控因子和motif,是有怎样的网络关系呢?
拿的是一些缺氧相关的基因最后算出来的motif和相关转录因子。(具体怎么算,可以之后写一期)
前面算得有问题可以私信发我邮箱,我发你们练习数据。
 当你手上有这样motif和转录因子的对应关系,然后就可以一样做图了。
当你手上有这样motif和转录因子的对应关系,然后就可以一样做图了。
library(visNetwork)motifs <- unique(as.character(edges[,1]))genes <- unique(as.character(edges[,2]))nodes <- data.frame(id=c(motifs, genes),label=c(motifs, genes),title=c(motifs, genes),shape=c(rep("diamond", length(motifs)), rep("elypse", length(genes))),color=c(rep("purple", length(motifs)), rep("skyblue", length(genes))))
这里的id是不能相同的,label是标签,title就是展示在图上的。后面可以按照自己需求制定形状还有颜色。其实还能加上别的。
nodesnew <- data.frame(id=c(motifsnew, genesnew),label=c(motifsnew, genesnew),title=c(motifsnew, genesnew), # tooltipfont.size=c(rep(60,8), rep(15, 365)),group=c(rep("TF", 8), rep("gene", 365)),shape=c(rep("diamond", length(motifsnew)), rep("elypse", length(genesnew))),color=c(rep("purple", length(motifsnew)), rep("skyblue", length(genesnew))))visNetwork(nodesnew, edgesnew,height = "1000px", width = "100px") %>%visOptions(highlightNearest = TRUE,nodesIdSelection = TRUE,manipulation = T) %>%visGroups(groupname = "gene", color = "skyblue") %>%visGroups(groupname = "TF", color = "purple",font.size=50,size = 50)
最后就能输出网页了。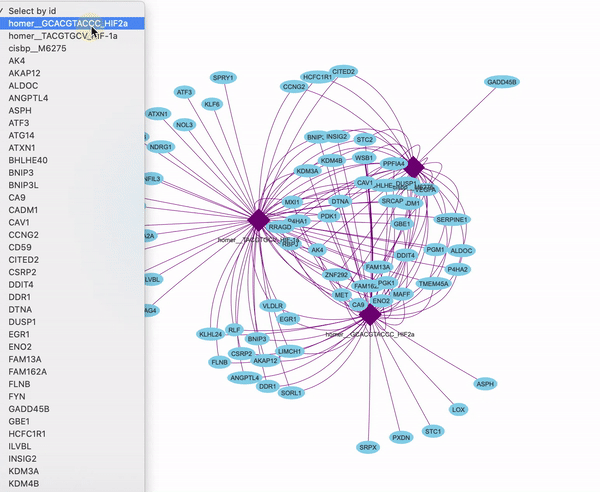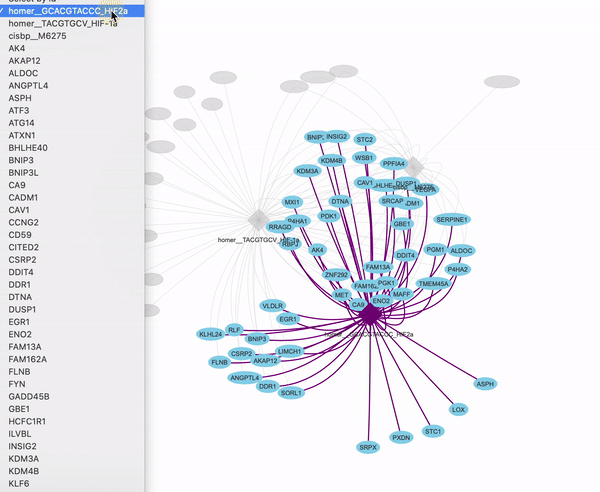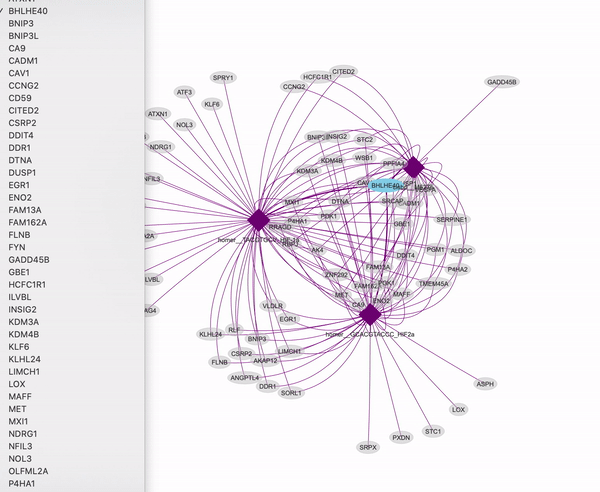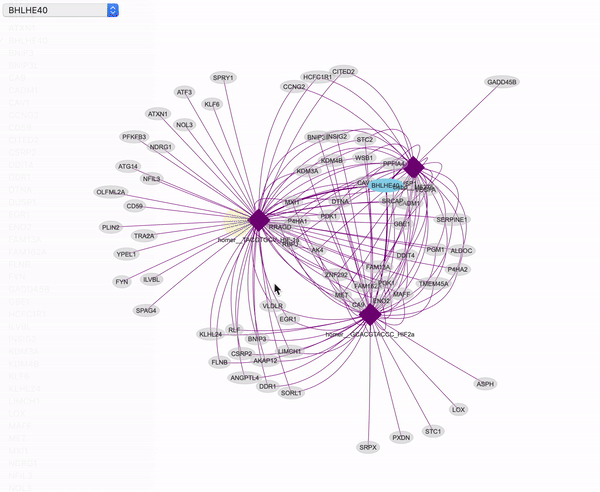
其实单细胞测序的分析,拿SCENIC做出来的东西,也一样能做成网络图。有兴趣的也可以讲。比如《Toward Minimal Residual Disease-Directed Therapy in Melanoma》这篇文章的Figur 6D的部分,就可以一样做。有需要的在写一期。
